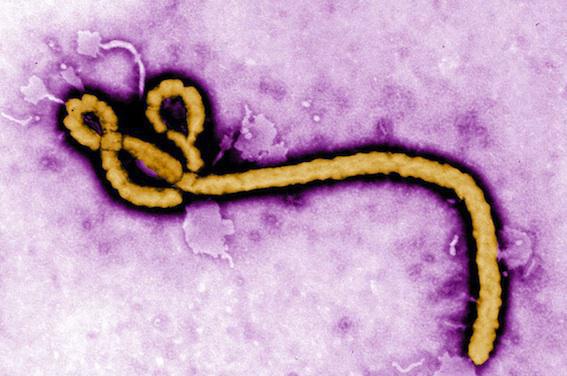
Plus d’un an après le début de l’épidémie d’Ebola en Afrique de l’Ouest, des chercheurs français de l’Institut Pasteur et du Centre national de la recherche scientifique (CNRS) (1) ont identifié 3 lignées du virus en Guinée. Cette découverte est primordiale pour mieux comprendre comment l’épidémie s’est propagée. Un véritable travail d’enquête publié ce jeudi dans la prestigieuse revue Nature.
Les chercheurs ont analysé 85 échantillons de sérum ou de salive de personnes infectées entre juillet 2014 et novembre 2014 en Guinée. Et ils ont finalement identifié 3 variants bien distincts du virus, leur permettant de combler les pièces manquantes du puzzle.
Retracer la propagation du virus
L’épidémie d’Ebola a démarré dans un petit village guinéen, dans une région forestière de la préfecture de Guéckédou. Quelques semaines plus tard, le virus s’est diffusé dans la capitale, Conakry, et dans les pays voisins comme la Sierra Leone, le Libéria, le Nigéria, le Sénégal et le Mali. « A ce moment-là, deux variants du virus Ebola ont suivi la route menant à la région très urbanisée de Conakry, explique Etienne Simon-Loriere, chercheur en biologie évolutive à l’Institut Pasteur et auteur de ces travaux. Puis, au début de l’été, un troisième variant qui a évolué en Sierra Leone est revenu dans la capitale ». L’une de ces lignées représenterait le chaînon manquant pouvant expliquer les deux introductions d’Ebola au Mali, en octobre, puis en novembre 2014.

Etienne Simon-Loriere, chercheur en biologie évolutive à l'Institut Pasteur (Paris) : « On a identifié une personne qui aurait fait le trajet de Sierra Leone à Conakry. C’est un exemple illustrant le challenge pour les personnes en charge de maîtriser une épidémie. »
Ainsi, tout laisse penser qu’à l’origine de l’épidémie, l’animal (sûrement la chauve-souris) a transmis à l’homme plusieurs variants du virus qui ont ensuite évolué chez l'homme, puis qu’une troisième ait émergé chez l’homme. Tous ces variants sont retrouvés jusqu’à la fin de l’été et bien au-delà. Une épidémie qui a beaucoup surpris le monde scientifique, car le virus n’avait jamais circulé de façon prolongée chez l’homme. « D’habitude, ce sont des petites épidémies qui s’éteignent très rapidement, mais là, le virus s’est répliqué pendant plus d’un an chez l’homme uniquement. L’analyse du génome du virus Ebola permettra de comprendre comment il s’adapte et évolue en dehors de son réservoir naturel », commente Etienne Simon-Loriere.
Une co-circulation des lignées
Selon leurs observations, les séquences génétiques des trois variants sont relativement proches, mais elles portent suffisamment de mutations, notamment au niveau de l’enveloppe virale, pour conférer aux virus de nouvelles propriétés. « Mais heureusement, toutes les données épidémiologiques montrent qu’il n’y a pas de différences entre ces 3 lignées en termes d’infectiosité, de transmission ou de mortalité », rassure le chercheur. Les outils de diagnostic utilisés sur le terrain sont toujours pertinents, ainsi que les candidats thérapeutiques et les vaccins en développement ».

Les Français ne sont bien sûr pas les seuls à s’intéresser au parcours du virus. Des chercheurs allemands et une équipe américaine ont eux aussi analysé des centaines de séquences génétiques à partir de prélèvements réalisés en Sierra Leone et en Guinée forestière. Des analyses confirmées par la publication française. Au total, les scientifiques disposent de plus de 1 000 séquences pour caractériser le virus. « Et toutes ces séquences semblent descendre les unes des autres, ce qui signifie que, pendant cette épidémie, il n’y a pas eu de transfert additionnel depuis l’animal vers l’homme. Cette transmission est donc un événement rare », explique le chercheur.
Plus de 11 000 morts
Il est malgré tout essentiel d’explorer ces différences pour être prêt à faire face à une prochaine épidémie, qui pourrait se reproduire, selon toutes les statistiques. Par ailleurs, cette diversité génétique peut également ouvrir de nouvelles voies thérapeutiques.
Depuis le début de l'épidémie, 27 341 cas ont été déclarés, dont 11 184 sont morts. Au Libéria, l’un des pays les plus touchés, le territoire a été déclaré indemne de la maladie il y a peu de temps. Mais le virus continue à faire des ravages en Afrique de l'Ouest. Si de nombreuses recherches ont été porteuses de d’espoirs, la vigilance doit toujours être de mise dans cette partie du globe.
(1) Les chercheurs français ont également collaboré avec l'Institut Pasteur de Dakar et l'université de Sydney (Australie)